11 Lectura del genoma
Durante los primeros años de la década de 1970, la ciencia desentrañó la estructura en doble hélice del ácido desoxirribonucleico (ADN), los tripletes que codifica para producir proteínas y muchas de las secuencias de aminoácidos que dan lugar a las proteínas. Hamilton Smith, Paul Berg y Herbert Boyer también habían dado los primeros pasos en el campo de la ingeniería genética, demostrando cómo pueden transferirse de un organismo a otro pequeños segmentos de ADN.
Sin embargo, los avances en la genética y sus beneficios para la medicina estaban dificultados por una barrera técnica considerable. Era extremadamente difícil entender qué segmentos de ADN actuaban como genes bien definidos y también el orden en el que estaban escrita las «letras» del ADN.
En 1969, el genetista norteamericano Jonathan Beckwith aisló el primer gen a partir de una bacteria, y el biólogo molecular belga Walter Fiers determinó en 1972 la primera secuencia de un gen (el gen de la cubierta proteica de un virus). Sin embargo, estos logros se consideraron mediante la lectura de copias del ácido ribonucleico (ARN) en el código genético, no a través de la lectura del ADN en sí mismo. La técnica era lenta e ineficaz y, dado que el ARN tiene un ciclo vital muy corto, sólo resultaba adecuada para los genes más pequeños. No había ningún método para leer de manera sistemática el orden de las bases del ADN y, así, las perspectivas de cartografiar los genes complejos —y no digamos las secuencias genéticas complejas— de los organismos grandes eran escasas.
Premios Nobel
Solamente hay cuatro personas que hayan recibido el premio Nobel en dos ocasiones, y en dos de ellas el galardón vino de la mano de sus descubrimientos en genética. Fred Sanger recibió el premio Nobel de química en dos ocasiones, y Linus Pauling recibió el de química y el de la paz. El premio Nobel de fisiología o medicina también ha estado dominado por la genética, sobre todo desde la década de 1950, cuando esta ciencia empezó a avanzar. La lista de los galardonados es una especie de «quién es quién» en la historia de la genética: Morgan, Muller, Beadle, Tatum, Crick, Watson, Wilkins, Nirenberg, Monod, Smith, Baltimore y Cohen. Cinco de los últimos siete premios también han sido otorgados a científicos que efectuaron descubrimientos relacionados con la genética.
Finalmente, Fred Sanger (un bioquímico británico que ya había recibido el premio Nobel por determinar la secuencia de aminoácidos de la insulina) desarrolló en 1975 un mejor método de secuenciación. Este avance dio lugar a un cambio completo de la biología, transformando las perspectivas para conocer y manipular las funciones de los genes y, en última instancia, permitiendo a los científicos cartografiar el código genético del ser humano.
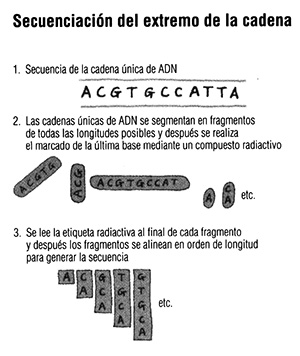
Secuenciación del genoma La novedad de la estrategia de Sanger consistió en el uso de una única cadena de ADN como plantilla para realizar cuatro experimentos en distintas placas de laboratorio. En cada una de ellas dispuso un caldo con las cuatro bases (A, C, G y T) y polimerasa del ADN, una enzima que actúa sobre las bases para producir una nueva cadena complementaria. Después, añadió a cada experimento un «ingrediente mágico»: una versión modificada de una de las bases que interrumpía la reacción tan pronto como actuaba sobre la cadena; Sanger marcó el extremo de la cadena con un compuesto radiactivo.
A medida que tiene lugar la reacción, se generan miles de fragmentos de ADN de longitudes variables, algunos de los cuales finalizan en todas las posiciones posibles. Después, estos fragmentos se introducen a través de un gel para separarlos por orden de longitud, de manera que el marcador radiactivo permite leer la base que está en el extremo de cada fragmento.
Si los primeros fragmentos, de sólo una base, muestran timina en el extremo, la primera letra es una T. Si los fragmentos de dos bases presentan citosina en el extremo, el código hasta el momento es TC. Los fragmentos de tres fases con guanina en el extremo hacen que la secuencia sea TCG. Después, cada fragmento se lee de la misma forma, hasta que todas las casillas del código se han rellenado con una letra.
Este sistema, denominado secuenciación del extremo de la cadena, era mucho más rápido que los métodos alternativos. Era eficaz, fiable y seguro; las demás técnicas que se desarrollaron durante la misma época requerían el uso de mayores cantidades de radiactividad y de productos químicos tóxicos. Este tipo de secuenciación se convirtió rápidamente en el método de elección para la lectura de los genes.
En los primeros tiempos, la técnica era manual. Cuando Sanger la utilizó para leer el genoma de un virus fago denominado Phi-X174 (el primer organismo cuyo ADN fue secuenciado en su totalidad), contó las bases una por una a partir de las bandas que aparecían en el gel. Obviamente, este proceso requería mucho tiempo y era costoso, pero también susceptible de automatización. En 1986, Leroy Hood (del California Institute of Technology) diseñó la primera máquina de secuenciación del ADN. En vez de utilizar radiactividad para identificar las bases, Hood las marcó con cuatro colorantes fluorescentes que brillaban cuando se escaneaban con un láser. Cada señal luminosa era identificada después por un ordenador, lo que permitía determinar la secuencia de una forma constante. No era necesario que los técnicos evaluaran los portaobjetos. Para la secuenciación del genoma humano se utilizaron los dispositivos de Applied Biosystems (la compañía que comercializó el invento de Hood).
Descubrimiento de genes Estas nuevas técnicas de secuenciación simplificaron la lectura de las letras que constituyen los genes. Para ello, los científicos tenían que purificar primero una proteína (como la adrenalina celular) y después determinar su secuencia de aminoácidos y todas las posibles combinaciones de tripletes de ADN en las que podrían estar escritas las instrucciones genéticas. El proceso podía requerir años.
A partir de estas secuencias candidato, era posible construir una «sonda de ADN» para localizar las secuencias en los cromosomas aprovechando una de las características de la doble hélice descubierta por Crick y Watson. Las cadena únicas de ADN se unían a otras cadenas únicas constituidas por bases complementarias; es decir, una secuencia ACGT se unía a una secuencia TGCA. Era posible marcar radiactivamente una sonda de ADN con parte de la secuencia del gen candidato y, después, mezclarla con el material genético procedente de los cromosomas. Si la sonda detectaba algo, posiblemente era el gen real que, después, se podía aislar, leer y cartografiar para determinar su posición en el cromosoma.
Con este método, a finales de la década de 1980 ya se habían localizado y secuenciado 2.000 genes. Uno de ellos era el que codifica la eritropoyetina, una proteína que estimula la producción de hematíes. Cuando la compañía Amgen desarrolló una versión recombinante de la eritropoyetina, el producto se convirtió en un medicamento que transformó radicalmente el tratamiento de la anemia. Sin embargo, a pesar de las grandes sumas invertidas por la industria farmacéutica, el ritmo de los descubrimientos siguió siendo bajo.
A principios de la década de 1990, esta velocidad se incrementó de manera súbita gracias a una nueva técnica de localización de genes diseñada por Craig Venter, un surfista californiano que se había iniciado tarde en la biología tras haber trabajado como auxiliar médico en Vietnam. Venter se dio cuenta de que la secuenciación de fragmentos pequeños de ADN que se copian en el ARN mensajero (la señal química que da lugar a la producción de las proteínas) permitía crear «etiquetas de secuencia expresada» que podrían facilitar la localización de genes enteros en el ADN cromosómico. Mediante este método, su laboratorio fue capaz de descubrir en poco tiempo hasta 60 nuevos genes al día. El genoma estaba empezando a revelar sus secretos.
El primer proyecto del genoma humano: el ADN mitocondrial
El genoma humano tiene una longitud de 3.000 millones de bases, y su lectura quedaba completamente fuera del alcance de las herramientas de secuenciación que utilizó Sanger a finales de la década de 1970. Sin embargo, ello no le impidió iniciar una especie de proyecto del genoma humano más modesto. Aunque la mayor parte del ADN humano se localiza en los cromosomas del núcleo celular, una pequeña cantidad permanece en las mitocondrias, estructuras productoras de energía. El equipo de Sanger secuenció este fragmento, más manejable, del código genético humano y en 1981 publicó los detalles de sus 16.569 bases y sus 37 genes.
Las mitocondrias son estructuras pequeñas, pero de gran importancia. Las alteraciones en los genes mitocondriales causan enfermedades en la actualidad, los científicos están investigando la forma de trasplantar las mitocondrias entre distintos óvulos para evitar la transmisión hereditaria de dichas enfermedades. Dado que las mitocondrias se transmiten por la línea femenina prácticamente sin modificaciones, el ADN que contienen también resulta útil para el estudio de la evolución y la genealogía.
Cronología:
1972: Walter Fiers (nacido en 1931) determina la primera secuencia genética
1975: Fred Sanger (nacido en 1918) desarrolla la técnica de secuenciación del extremo de la cadena
1977: Sanger secuencia el primer genoma de un organismo completo, un virus fago denominado Phi-X174
1981: El equipo de Sanger secuencia el genoma mitocondrial humano
1991: Craig Venter (nacido en 1946) desarrolla un nuevo método rápido para el descubrimiento de genes a través de etiquetas de secuencias expresadas
La idea en síntesis: los genes pueden ser aislados y leídos